Creando diagramas Estratigraficos
som.RmdPrerequisitos
En este mini tutorial trabajaremos con los packages ggplot y tidypaleo “modificado del tutorial de Dewey Dunnington (2018)” para hacer una figura en profundidad de datos obtenidos por Romina en conchales.
Instalación
Active o desactive # para instalar algún package que necesitemos
# Desde CRAN
# install.packages("ggplot2")
# install.packages("tidyverse")
# install.packages("here")
# Desde Github
#devtools::install_github("thomasp85/patchwork")
#devtools::install_github("paleolimbot/tidypaleo")Cargando los datos que necesitaremos
Primero cargamos los datos desde nuestra carpeta (recomiendo usar el package here para que podamos reproducir las figuras desde cualquier maquina sin cambiar el directorio)
mnidata <- read_csv(here::here("data-raw/MNI.csv"))Analizando los datos NMI y NISP.
Datos NMI
Primero debemos ordenar los datos para que puean ser leido por ggplot, para esto usamos las herraminetas disponibles en el tidyverse package con la función gather(), la idea es dejar todos los valores en una sola variable que se conoce como tabla larga.
## reordenamos los datos en un formato de tabla larga
mnidata<- gather(mnidata, key = species, value = mni, 4:17)
mni_plot <- ggplot(mnidata, aes(x = mni, y = depth)) +
geom_lineh() +
geom_point() +
scale_y_reverse() +
facet_geochem_gridh(vars(species)) +
labs(x = "MNI", y = "Depth (cm)")
## Una forma de resaltar un rango de profundidades
mni_plot 
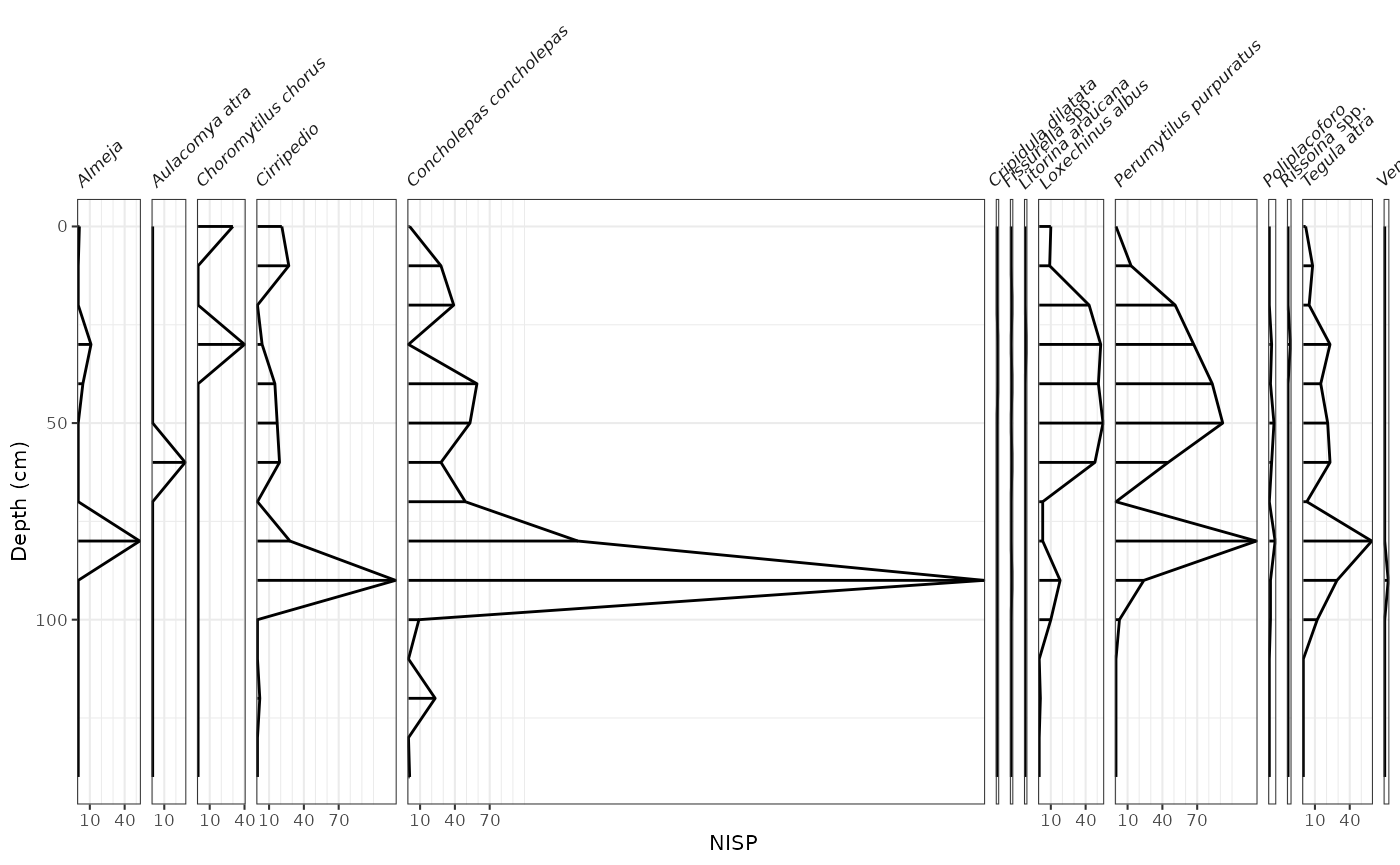
## O simplemente graficamos con lineas y puntos
mni_ggplot <- ggplot(mnidata, aes(x = mni, y = depth)) +
geom_col_segsh() +
geom_lineh() +
scale_y_reverse() +
facet_abundanceh(vars(species)) +
labs(x = "MNI", y = "Depth (cm)")
mni_ggplot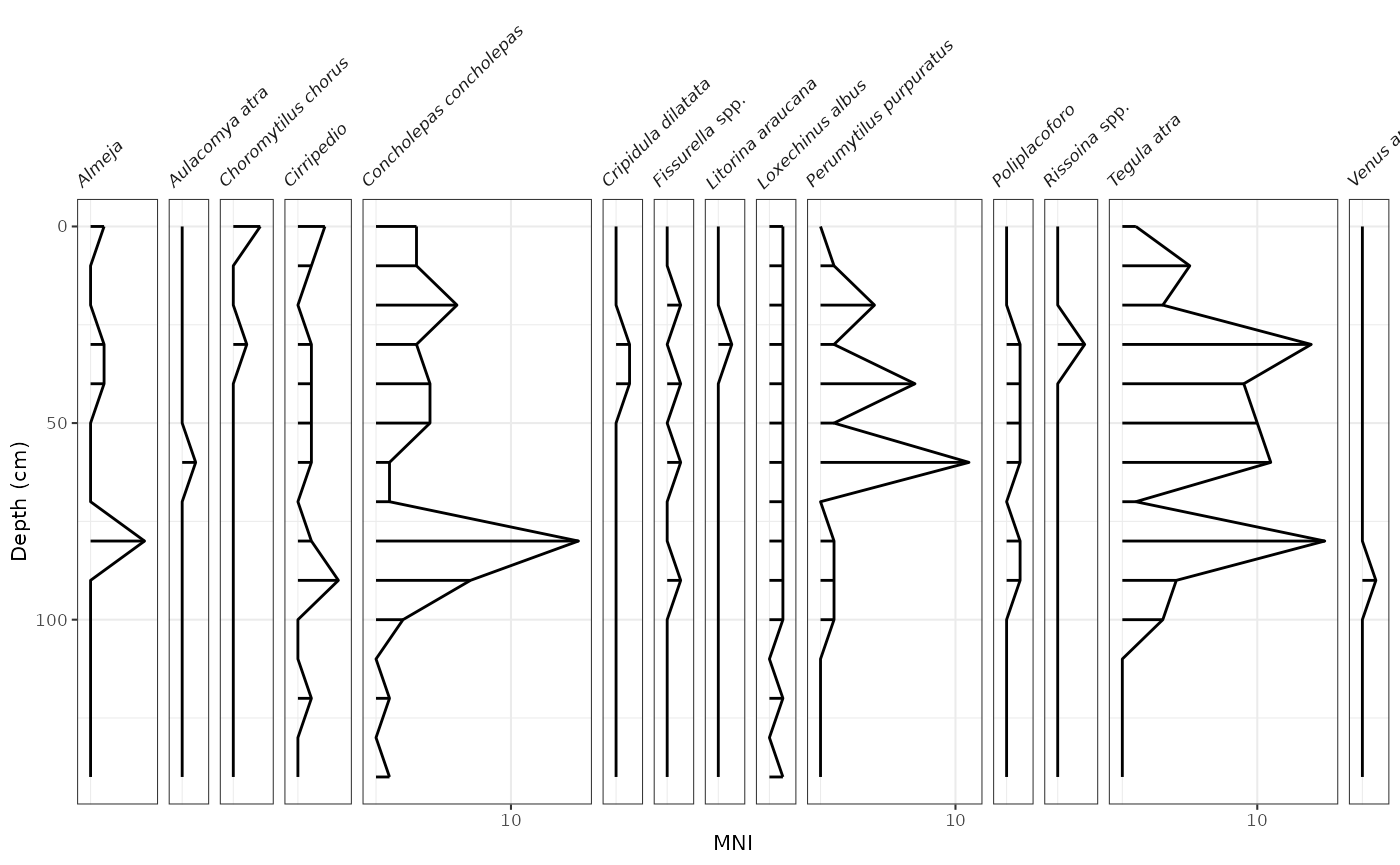
Datos NISP
Ahora cargamos los datos de NMI y repetimos las lineas de arriba, cambiando el nombre del archivo por
nispdata <- read_csv(here::here("data-raw/NISP.csv"))Ordenamos los datos en un formato de tabla larga
## reordenamos los datos en un formato de tabla larga
nispdata<- gather(nispdata, key = species, value = nisp, 4:17)
## Y graficamos con ggplot
nisp_plot <- ggplot(nispdata, aes(x = nisp, y = depth)) +
geom_lineh() +
geom_point() +
scale_y_reverse() +
facet_geochem_gridh(vars(species)) +
labs(x = "NISP", y = "Depth (cm)")
## Una forma de resaltar un rango de profundidades
nisp_plot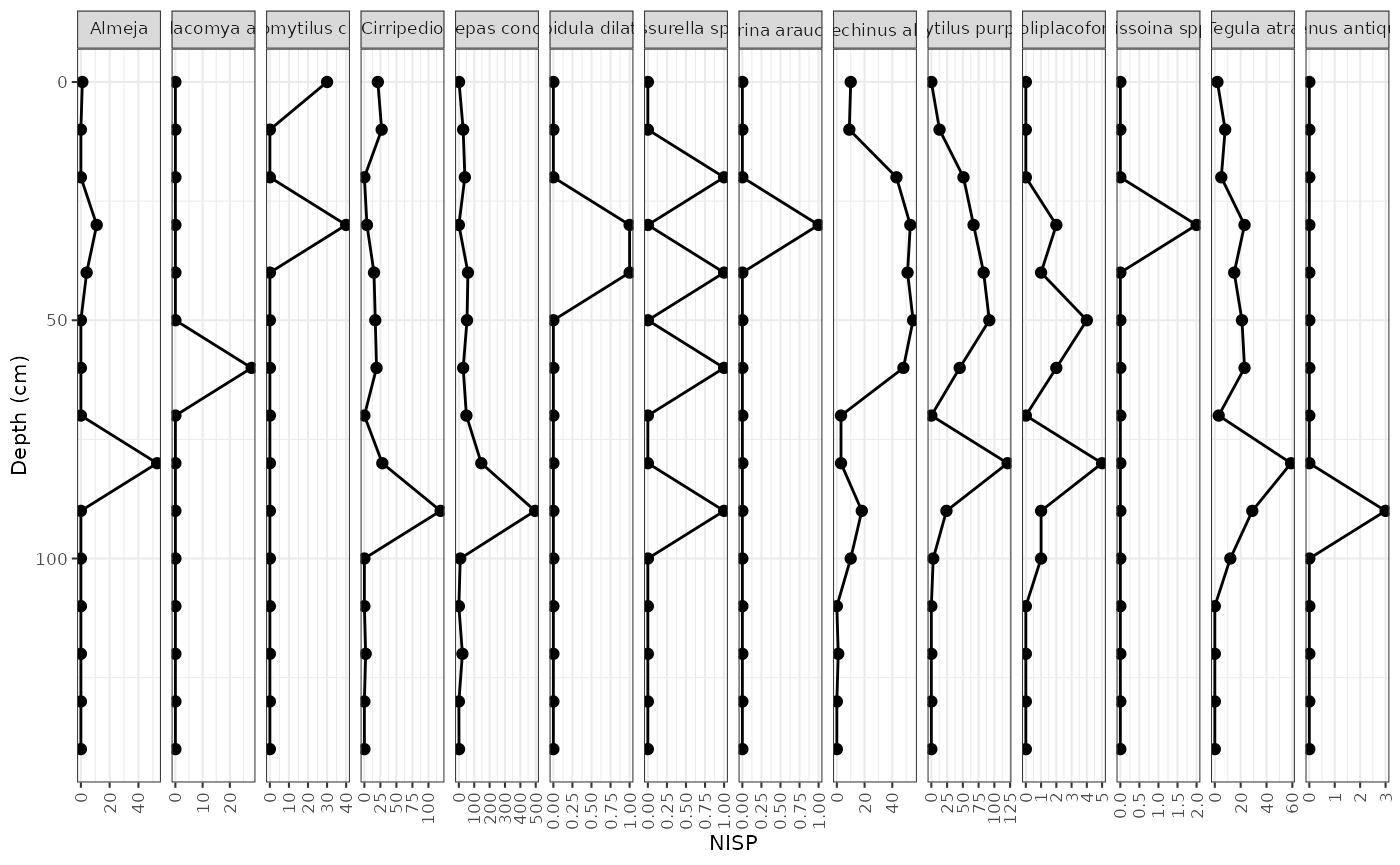
## O simplemente graficamos con lineas y puntos
nisp_ggplot <- ggplot(nispdata, aes(x = nisp, y = depth)) +
geom_col_segsh() +
geom_lineh() +
scale_y_reverse() +
facet_abundanceh(vars(species)) +
labs(x = "NISP", y = "Depth (cm)")
nisp_ggplot